4 Classificação Taxonômica
4.1 Contexto
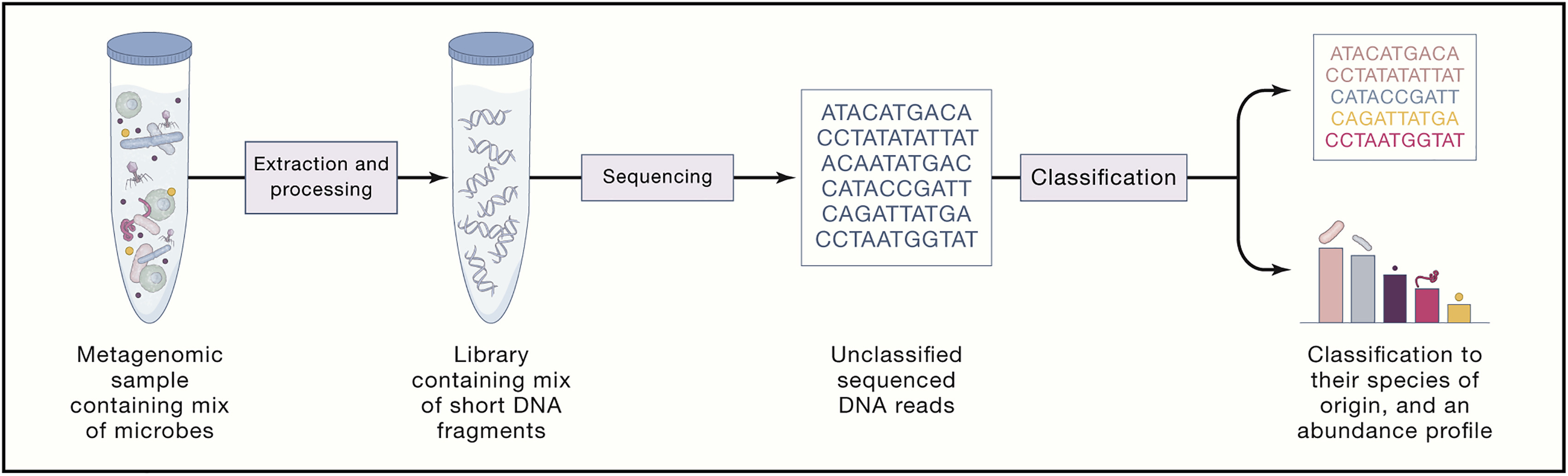
O passo de classificação taxonômica trata de classificar as sequências mistas, sejam leituras ou contíguas, de uma amostra metagenômica em táxons claramente definidos, e tipicamente acompanha um perfil de abundância desses táxons.
4.2 Adquirindo o banco de dados
Mude o diretório atual para "Pipeline/alignment/db" (pois esse banco também será usado na seção Seção 5.2.1, do alinhamento) e baixe o banco de dados de proteínas do NCBI-nr:
O banco de dados é muito grande e inviável de se instalar em um computador pessoal. Use um cluster de alta performance ou ambientes similares!
wget ftp://ftp.ncbi.nlm.nih.gov/blast/db/FASTA/nr.gz
pigz -d nr.gz -p 8Construa um índice DIAMOND:
diamond makedb --in nr -d ../index/nrMude o diretório atual para “Pipeline/taxonomic/db” e baixe os seguintes arquivos:
wget ftp://ftp.ncbi.nlm.nih.gov/pub/taxonomy/taxdump.tar.gz
wget ftp://ftp.ncbi.nlm.nih.gov/pub/taxonomy/accession2taxid/prot.accession2taxid.gzEsses são os bancos de dados que utilizaremos para executar o Kaiju
Extraia e descompacte os arquivos necessários:
tar -xf taxdump.tar.gz nodes.dmp names.dmp merged.dmp
pigz -d prot.accession2taxid.gz -p 8Construa um índice Kaiju:
kaiju-convertNR -t nodes.dmp -g prot.accession2taxid -m merged.dmp -e ~/miniconda3/envs/medusaPipeline/bin/kaiju-excluded-accessions.txt -a -o kaijuNR.fasta -i ../../alignment/db/nr
kaiju-mkbwt -n 8 -a ACDEFGHIKLMNPQRSTVWY -o kaijuNR kaijuNR.fasta
kaiju-mkfmi kaijuNRNa chamada kaiju-convertNR, por padrão, são incluídas apenas sequências de Archaea, Bactérias e Vírus do NCBI-nr. Este comportamento pode ser alterado com o argumento -l, passando um arquivo de entrada como ~/miniconda3/envs/medusaPipeline/bin/kaiju-taxonlistEuk.tsv. Este argumento utiliza apenas sequências com ancestrais listados no arquivo.
4.3 Realizando a classificação taxonômica
Mude o diretório atual para “Pipeline/taxonomic” e execute a classificação taxonômica:
kaiju -t db/nodes.dmp -f db/kaijuNR.fmi -i ../data/removal/unaligned_1.fastq -j ../data/removal/unaligned_2.fastq -o ../result/SRR579292_kaiju.out -z 8Adicione os nomes dos táxons ao output:
kaiju-addTaxonNames -t db/nodes.dmp -n db/names.dmp -r superkingdom,phylum,class,order,family,genus,species -i ../result/SRR579292_kaiju.out -o ../result/SRR579292_kaiju.namesGere os gráficos Krona:
kaiju2krona -t db/nodes.dmp -n db/names.dmp -i ../result/SRR579292_kaiju.out -o ../result/SRR579292_kaiju2krona.out
ktImportText -o ../result/SRR579292_krona.html ../result/SRR579292_kaiju2krona.out